近日,国际著名学术期刊Nucleic Acids Research《核酸研究》发表了我校生命科学学院戴雄风/朱曼璐团队在合成生物学领域的最新进展,论文题目为“Systematic modulation of bacterial resource allocation by perturbing RNA polymerase availability via synthetic transcriptional switches”(https://doi.org/10.1093/nar/gkaf814)。朱曼璐教授为该论文的第一兼通讯作者,戴雄风教授为该论文的通讯作者,我校为该论文的独立完成单位。
细菌是最常用的合成生物学底盘系统之一。生长繁殖作为细菌底盘系统的核心天然功能,需要细菌集中胞内大部分资源包括碳氮代谢物、能量、核糖体等进行支撑,因而与底盘细菌的生理代谢状态息息相关,进而会直接影响人工合成回路的运行效率。因此,研究细菌生长调控机制对于指导合成生物学理性设计意义重大,不仅涉及优化底盘系统的生长代谢性能,也关乎提高合成回路与底盘系统的生理适配性。
目前合成生物学领域与代谢工程领域一个重要的瓶颈问题在于:底盘细菌生长与人工合成回路的权衡(Nat Rev Bioeng 2024;2:1039–55)。这一挑战的核心在于细胞资源的有限性——当引入外源合成途径时,底盘系统必须在维持自身生长和完成外源目标产物合成之间分配资源(如能量、核糖体、前体代谢物等)。当宿主优先保障自身生长代谢状态时,往往会导致外源合成效率低下。因此,设计创制高效的生长-生物合成的动态平衡调控体系对于合成生物学、代谢工程实践至关重要。
在最新的研究中,该团队以细菌RNA聚合酶为调控靶点,在芽孢杆菌中构建了两类转录开关:(1)看家σ因子(SigA)扰动系统:通过精确控制细菌管家σ因子的表达,改变不同类型RNA聚合酶的分布。(2)核心酶(RpoBC)扰动系统:靶向RNA聚合酶核心亚基ββ'的合成,影响RNA聚合酶的整体浓度。实验表明,这两个正交转录开关都可以通过对RNA聚合酶的浓度或者分布的调控,实现了对细菌生长代谢状态的精细调控。
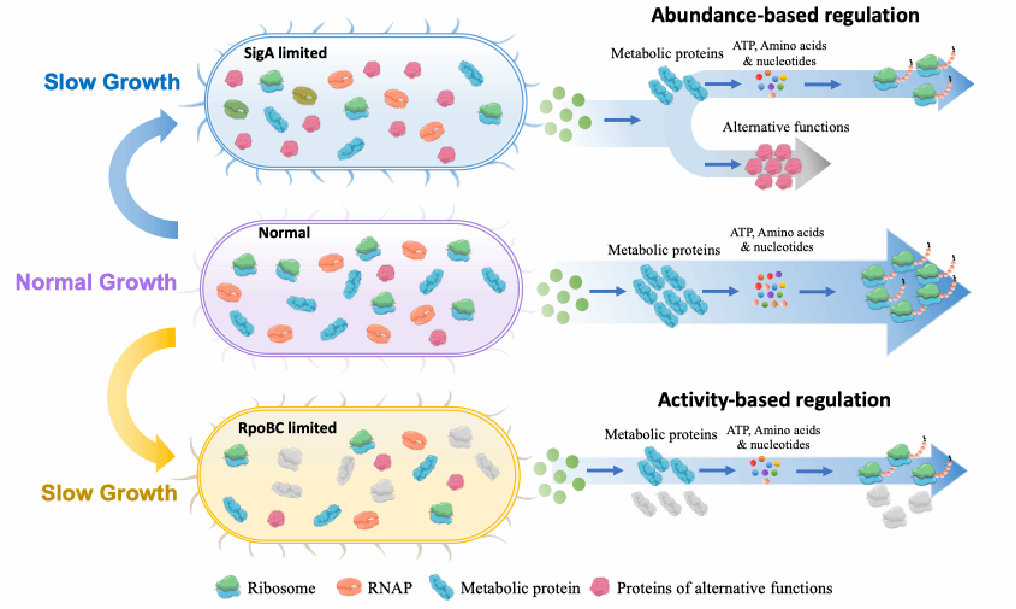
图1:两类全局转录开关:(1)σ因子(SigA)调控系统(2)核心酶(RpoBC)调控系统,对细菌胞内资源配置的调控规则。
进一步地,研究团队通过多组学分析(转录组、蛋白质组、多聚核糖体分析等)发现:SigA限制触发“丰度调控”:降低SigA表达后,细菌将资源从生物合成途径(如氨基酸代谢)转向应激响应通路,形成“生长-生产”动态切换。这种重分配效应可用于优化生物制造效率,例如提高目标蛋白质的产量。而RpoBC耗尽引发“活性调控”:核心酶不足导致核糖体翻译起始受阻而失去活性;此外还激活DNA损伤修复系统(如RecA、UvrBA通路),显著提升细菌对紫外线或丝裂霉素的敏感性。这一发现为抗生素增效剂设计提供了新靶点。
总体而言,该团队通过开发合成转录开关调控RNA聚合酶的可用性,揭示了细菌资源分配的全新机制,为合成生物学在生物制造和医疗领域的应用提供了理论基石。
该团队近年来在定量与合成生物学领域还取得了以下研究进展:揭示了模式细菌生长与基因表达的全局调控机制,阐释了细菌生长调控的权衡规则,突破了“生长最大化”主流理论(Nature Communications. 2024a,15(1): 4238; Science Advances. 2023, 9(39): eadg9733; Nature Communications. 2023, 14(1):467);揭示了细菌生长与转录翻译速度的全新耦合机制,改写了“核糖体推行RNA 聚合酶”主流模型(PNAS, 2025, 122 (18): e2427091122; Nat Microbiol, 2019, 4(12), 2347-2356;Sci. China Life Sci, 2021, 64(5), 795–802; Trends Biochem Sci, 2020, 45(8): 681-692);揭示了基因表达时效性对于细菌适应生长逆境以维持种群适合度的关键意义(Nature Communications, 2024b, 15: 9567)。系列成果为理解细菌复杂的生理调节网络及其顶层设计原理开辟了新视野,并深化了学界对微生物适应性进化机理的认知。
该工作得到了国家重点研发计划(青年科学家项目)、国家自然科学基金、武汉市科技重大专项、华中师范大学“学者名师支持计划”以及中央高校基本科研业务费的资助。
文字来源: 戴雄风、朱曼璐课题组
图片来源:戴雄风、朱曼璐课题组
初审初校:李韧
复审复校:万翠红
终审终校:邱保胜